Table of Contents
PG 552: Drug Hunting
Navigation und visuelle Analyse im pharmakologischen Strukturraum
Neuigkeiten
- E-Mail-Verteiler:
Termine
Abschlussvortrag
Donnerstag, 13.10.2011, 10:15 Uhr, OH14 Raum 202
Seminarphase
- Dienstag, 12.10, 14:00 Uhr, OH14 Raum 202
- Donnerstag, 14.10, 14:00 Uhr, OH14 Raum 202
- Dienstag, 19.10, 14:00 Uhr, OH14 Raum 202
- Donnerstag, 21.10, 14:00 Uhr, OH14 Raum 202
Vorbesprechung
Freitag, 16.07, 10:15 Uhr, OH14 Raum 202
Einzelpräsentation
Dienstag, 18.05, 10:15 Uhr, OH14 Raum 202
Teilnehmer
- Bernhard Dick
- Thorsten Flügel
- Henning Garus
- Michael Hesse
- Philipp Kopp
- Philipp Lewe
- Dominic Sacré
- Till Schäfer
- Thomas Schmitz
- Ömer Uzun
Organisation
Zeitraum
WiSe 2010/11, SoSe 2011
Umfang
8 SWS im ersten und zweiten Semester, insgesamt 16 SWS
Veranstalter
Kooperationspartner
- Prof. Dr. Herbert Waldmann, Max-Planck-Institut für molekulare Physiologie, Dortmund
Thema
Entwicklung einer Java-basierten Software zur Navigation und visuellen Analyse im pharmakologischen Strukturraum mit Hilfe von Verfahren zur Visualisierung und Klassifikation biochemischer Daten.
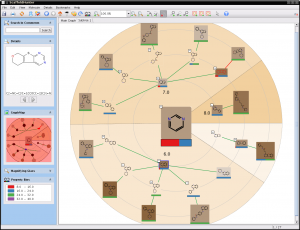 |  |
| Scaffold Hunter | Molwind |
| Software zur Visualisierung des chemischen Strukturraums | |
Motivation
Das Finden neuer Wirkstoffe mit pharmazeutischem Nutzen ist eine Kernaufgabe der chemischen Biologie. Innerhalb des chemischen Strukturraums (der Gesamtheit aller theoretisch möglichen Moleküle) kommt nur ein geringer Teil von kleinen, biologisch aktiven Molekülen als Wirkstoff in Frage. Die geschätzte Anzahl möglicher Wirkstoffkandidaten in diesem pharmakologischen Strukturraum ist mit 10^60 jedoch noch immer immens, so dass eine Suche nach neuen Wirkstoffen gezielt erfolgen muss und sich auf einen möglichst kleinen, relevanten Teil beschränken sollte. Entsprechende Datensätze von chemischen Strukturen werden in sogenannten Substanzbibliotheken zusammengefasst und können experimentell in Hochdurchsatzverfahren auf ihre Eignung als Wirkstoff getestet werden.
Auch wenn einige Kriterien für eine automatisierte Vorauswahl herangezogen werden können, setzt die Bewertung der biologischen Wirksamkeit zur Auswahl von Wirkstoffkandidaten Expertenwissen über komplexe biochemische Zusammenhänge voraus. Dazu wird eine möglichst vollständige Übersicht der schon bekannten Eigenschaften einer Substanz sowie deren bestehender Ähnlichkeiten zu bereits untersuchten Molekülen benötigt. Die Integration und geeignete visuelle Aufbereitung der zur Verfügung stehenden Daten im Rahmen von Softwaretools kann den Analyseprozess für den Experten wesentlich vereinfachen und beschleunigen. Konzepte zur sinnvollen Ordnung des Strukturraums und ihre graphische Visualisierung sind dabei ein geeignetes Hilfsmittel. Waldmann et al. haben ein Konzept zur Klassifikation der Moleküle eines Datensatzes und ihre Einordnung in einen hierarchischen Strukturgerüstbaum entwickelt. Dabei werden Moleküle mittels Methoden der Chemoinformatik chemisch sinnvollen Strukturgerüsten, den sogenannten Scaffolds, zugeordnet, die wiederum schrittweise vereinfacht werden indem sukzessiv Ringe der Struktur entfernt werden. Hieraus ergibt sich eine Vielzahl von Elter-Kind-Beziehungen, die sich zu einem Baum zusammenfassen lassen.
Mit der Software Scaffold Hunter wurde bereits einmal auf Grundlage dieses Konzepts ein Navigationstool in einer Projektgruppe (PG504 ChemBioSpacE) erfolgreich umgesetzt. Der Scaffold Hunter erlaubt die interaktive Visualisierung eines Strukturgerüstbaum und dadurch die intuitive Navigation im chemischen Strukturraum. Hierbei wird die Baumstruktur zuvor nach einem festen Regelsatz berechnet und in einer Datenbank gespeichert. Einen vergleichbaren Ansatz verfolgt die Software Molwind, wobei die Visualisierung auf der Open Source Software NASA World Wind beruht und Methoden aus der Kartographie und der Geoinformatik zur Navigation im Strukturraum eingesetzt werden. Die positive Rückmeldung zu solchen Konzepten von Forschungsgruppen und der pharmazeutischen Industrie motiviert zu einer Weiterentwicklung der Ansätze.
Die Festlegung auf ein einziges Konzept zur Klassifikation des Strukturraums schränkt die Nutzbarkeit der Software jedoch ein. Einige Ordnungskonzepte erscheinen für bestimmte Fragestellungen optimal, unterstützen andere Anwendungsfälle jedoch möglicherweise nur unzureichend. Daraus resultiert der Wunsch nach einer flexiblen Ordnung des Strukturraums, die je nach Fragestellung gewählt werden kann.
Der Nutzen derartiger Software kann zudem wesentlich von der Verknüpfung der Moleküle mit zusätzlichen Informationen profitieren. Dazu zählen beispielsweise die chemischen Eigenschaften eines Moleküls, ihre Fähigkeit an bestimmte Proteine zu binden, die diesbezügliche Selektivität, ihre Verfügbarkeit sowie bekannte pharmazeutische Wirkungen. Sinnvolle Konzepte für das Knowledge Management, also Methodiken, die den möglichst effizienten Umgang mit diesen Daten erlauben, werden sowohl von der Forschung als auch der Industrie als extrem wichtig angesehen und bilden einen Pfeiler der von der EU und der pharmazeutischen Industrie zusammen angestoßenen Innovative Medicine Initiative. Auch wenn das Wissen über die Beziehung zwischen Eigenschaften der Moleküle und ihrer Wirkung teilweise noch lückenhaft ist, und zu einem großen Bereich des chemischen Strukturraums noch gar keine Informationen vorliegen, so ist doch bereits eine große Anzahl von Molekülen gut untersucht. Während derartige Daten lange Zeit schwer zugänglich waren, werden die resultierenden Ergebnisse mittlerweile systematisch in Datenbanken gespeichert und mit Projekten wie z.B. PubChem oder ChEBI stehen inzwischen zahlreiche freie, öffentliche Datenbanken zur Verfügung. Die für typische Fragestellungen benötigten Daten liegen aber unter Umständen verstreut vor und sind nicht gut untereinander verknüpft. Zusätzlich müssen üblicherweise sowohl lokale Datenbanken (mit noch vertraulichen Daten) und öffentlich zugängliche Datenbanken zusammen betrachtet werden. Die Schwierigkeit besteht derzeit also darin, relevante Informationen unterschiedlicher Datenbanken zusammenzuführen (Datenintegration), auszuwerten, auf angemessene Weise zu repräsentieren und dadurch erfassbar zu machen. Hier soll die Projektgruppe Lösungen entwickeln und umsetzen, die es zum Beispiel ermöglichen, Eigenschaften von Molekülen vorherzusagen, oder vielversprechende aber noch nicht synthetisierte Moleküle in der Substanzbibliothek zu finden.
Aufgabenstellung
Die wenigen bereits existierenden Navigationstools bieten keine Schnittstelle für die Einbindung von im Internet verfügbaren Wissensressourcen, sondern erlauben nur die Repräsentation der Daten aus einer einzelnen Datenbank. In der Projektgruppe soll ein Konzept entwickelt und umgesetzt werden, das es erlaubt, unterschiedliche Datenquellen in Software zur visuellen Analyse von chemischen Moleküldatenbanken zu integrieren. Dazu soll ein flexibles Framework erstellt werden, das es ermöglicht, Informationen aus frei verfügbaren Datenbanken automatisch mit chemischen Strukturen zu verknüpfen. Dies beinhaltet zum einen die technische Unterstützung der Einbindung durch entsprechende Schnittstellen und zum anderen die geeignete graphische Repräsentation. Der großen Vielfalt an möglichen Datenquellen soll dadurch Rechnung getragen werden, dass sich weitere Datenbanken mit geringem Aufwand anbinden lassen.
Zusätzlich bauen bestehende Tools auf einer statischen Umsetzung einer spezifischen Molekülklassifikation auf, die jeweils nur einen kleinen Bereich der möglichen Fragestellungen abdeckt. Um die aus den Datenquellen bezogenen Informationen sinnvoll bei der Suche nach Wirkstoffen einsetzen zu können, müssen sie einerseits angemessen visuell repräsentiert werden, und andererseits sinnvoll in die Navigationsmechanismen integriert werden. Während zum Beispiel bisher die Navigation über einen statisch generierten Strukturgerüstbaum erfolgt, der rein auf strukturell begründeten Eltern-Kind-Beziehungen beruht, kann die Einbindung zusätzlicher Daten zu einer flexiblen und benutzerdefinierte Erstellung von Molekülrelationen genutzt werden. Für eine erste Erweiterung kann der in Scaffold Hunter integrierte Ansatz verwendet werden, wobei Elter-Kind-Beziehungen auf unterschiedliche Weise nach benutzerdefinierten Regeln bestimmt werden. Darüber hinaus sollen statistische Verfahren wie die Clusteranalyse verwendet werden, um dynamisch eine Klassifikation der Strukturen zu berechnen. Die Ergebnisse müssen geeignet visualisiert werden und eine interaktive Navigation in den Daten soll unterstützt werden. Die Clusteranalyse soll auf Basis verschiedener Eigenschaften der chemischen Strukturen durchgeführt werden, die vom Nutzer spezifiziert werden. Dazu sollen Informationen unterschiedlicher Datenquellen gewählt werden können.
Bei der Umsetzung der Konzepte muss daran gedacht werden, dass die Software für Anwender aus den Bereichen Chemie und Biologie gedacht ist, und die Interaktion mit der Nutzeroberfläche möglichst intuitiv vonstatten gehen soll. Eine zusätzliche Herausforderung ist das Volumen der zu verarbeitenden Daten. Hier müssen ausreichend effiziente Algorithmen und Methoden gewählt und unter Berücksichtigung des Ressourcenverbrauchs entsprechend umgesetzt werden.
Anforderungen
Teilnahmevoraussetzungen
- Programmiererfahrung in Java (V)
- Mensch-Maschine-Interaktion oder Effiziente Algorithmen oder Datenvisualisierung oder Automatisches Zeichnen von Graphen (M)
- Webtechnologien oder Wissensentdeckung in Datenbanken (W)
Legende: (M) Mindestens eine; (V) Voraussetzung; (W) Wünschenswert
Es werden keine Vorkenntnisse aus der Chemie benötigt.
Minimalziele
- Entwurf und Implementierung eines Frameworks zur Integration von zusätzlichen Informationen, das mindestens zwei externe sowie eine lokale Datenquellen umfasst und die beispielhafte Integration in Scaffold Hunter.
- Verknüpfung der Informationen mit den Strukturen im Programm, angemessene Aufbereitung und Darstellung.
- Implementierung weiterer Konzepte der Klassifikation und Navigation im chemischen Strukturraum sowie ihre Visualisierung.
- Erweiterung des bestehenden Konzepts statischer Strukturgerüstbäume.
- Anwendung statistischer Methoden zur Klassifikation des Strukturraums.
Material
Literatur
Chemoinformatik, Grundlagen
- Chemoinformatics - an introduction for computer scientists.
Nathan Brown; ACM Computing Surveys, Volume 41, Issue 2 (February 2009). - Chemical space and biology.
Christopher M. Dobson; Nature, Volume 432, No. 7019. (15 December 2004), pp. 824-828.
Scaffold Tree Konzept
- The Scaffold Tree - Visualization of the Scaffold Universe by Hierarchical Scafold Classification.
Schuffenhauer, A., P. Ertl, S. Roggo, S. Wetzel, M. A. Koch und H. Waldmann; J. Chem. Inf. Modelling, 47(1):47–58, January 2007. - Staring off into chemical space.
Irwin, John J.; Nature Chemical Biology, 5(8):536–537, August 2009. - Bioactivity-guided mapping and navigation of chemical space.
Renner, Steffen, Willem A. L. van Otterlo, Marta Dominguez Seoane, Sabine Mocklinghoff, Bettina Hofmann, Stefan Wetzel, Ansgar Schuffenhauer, Peter Ertl, Tudor I. Oprea, Dieter Steinhilber, Luc Brunsveld, Daniel Rauh und Herbert Waldmann; Nature Chemical Biology, 5(8):585–592, June 2009. - A Scaffold-Tree-Merging Strategy for Prospective Bioactivity Annotation of gamma-Pyrones.
Wetzel, Stefan, Wolfram Wilk, Samy Chammaa, Bianca Sperl, Anke G. Roth, Aybike Yektaoglu, Steffen Renner, Thorsten Berg, Christoph Arenz, Athanassios Giannis, Tudor I. Oprea, Daniel Rauh, Markus Kaiser und Herbert Waldmann; Angewandte Chemie International Edition, 49(21):3666–3670, 2010. - Bioactivity-guided navigation of chemical space.
Bon, Robin S. und Herbert Waldmann; Accounts of Chemical Research, 2010, 43, 1103-1114
Visualisierung
- Interactive exploration of chemical space with Scaffold Hunter.
Wetzel, Stefan, Karsten Klein, Steffen Renner, Daniel Rauh, Tudor I. Oprea, Petra Mutzel und Herbert Waldmann; Nature Chemical Biology, 5(8):581–583, August 2009. - Kartographie der Moleküle.
C. Herhaus und O. Karch; Nachrichten aus der Chemie, 57(10):1002–1004, 2009. - iPHACE: integrative navigation in pharmacological space.
Ricard Garcia-Serna, Oleg Ursu, Tudor I. Oprea und Jordi Mestres; Bioinformatics, 2010 Apr 1;26(7):985-6. - Data visualization during the early stages of drug discovery.
Dharmesh Maniyar und Ian Nabney; J. Chem. Inf. Model., 2006, 46 (4), pp 1806–1818. - Merging chemical and biological space: Structural mapping of enzyme binding pocket space.
Nils Weskamp, Eyke Hüllermeier, Gerhard Klebe; Proteins. 2009 Aug 1;76(2):317-30. - Constructing Overview + Detail Dendrogram-Matrix Views.
Jin Chen, Alan M. MacEachren, Donna J. Peuquet; IEEE Transactions on Visualization and Computer Graphics, IEEE Computer Society, 2009, 15, 889-896
Datenintegration
- Data integration: challenges for drug discovery.
David B. Searls; Nature Reviews Drug Discovery 4, 45-58 (January 2005) - Integrating biological databases.
Lincoln D. Stein; Nature Reviews Genetics 4, 337-345 (May 2003). - Integration of Biological Sources: Current Systems and Challenges Ahead.
Thomas Hernandez, Subbarao Kambhampati; SIGMOD Rec., 33(3):51–60, 2004. - Informationsintegration: Architekturen und Methoden zur Integration verteilter und heterogener Datenquellen.
Ulf Leser, Felix Naumann; dpunkt, 2006. - Biana: a software framework for compiling biological interactions and analyzing networks.
García-García J, Guney E, Aragues R, Planas-Iglesias J, Oliva B; BMC Bioinformatics 2010, 11:56doi:10.1186/1471-2105-11-56.
Datenbanken
- PubChem: a public information system for analyzing bioactivities of small molecules.
Yanli Wang, Jewen Xiao, Tugba O. Suzek, Jian Zhang, Jiyao Wang, and Stephen H. Bryant; Nucl. Acids Res., 37:W623–633, July 2009. - ChEBI: a database and ontology for chemical entities of biological interest.
Kirill Degtyarenko, Paula de Matos, Marcus Ennis, Janna Hastings, Martin Zbinden, Alan Mcnaught, Rafael Alcántara, Michael Darsow, Mickaël Guedj, Michael Ashburner; Nucleic Acids Research, 36 (Database issue):D344–D350, January 2008.
